Last Update June 19th, 2014
目次
5.1.Chromosome Overviewメニューからの検索
5.2.Chromosome Detailsメニューからの検索
7.1.Chromosome Overviewメニューから遺伝子を検索する
7.2.Chromosome Detailsメニューから遺伝子を検索する
8.1.クラシファイ機能の利用1−変異の付加情報から変異を分類表示する
8.2.クラシファイ機能の利用2−変異の付加情報を絞り込んで変異を表示する
1. 目的
遺伝子病や癌などの遺伝子の変異により生じる疾患は4,300種以上報告されています。それらの原因遺伝子の追究がゲノム解析の一環として精力的に進められており、現在特定染色体にマップされたものは約1,400種、原因遺伝子の同定及び変異の実体まで明らかにされたものは約800種あります。しかし、このような遺伝子変異データは、個々の研究者が様々な形式及びソフトウェアを用いて独立に蓄積しているのが現状です。また、それらの大多数は塩基配列変化等の文字データを蓄積したものに過ぎません。遺伝子変異データの効率化、体系的利用のためには、それらを統一し、同じ利用環境で使えるようにするとともに、OMIM、GDB、配列データベース等も利用して、データ項目間を有機的に結んだビジュアル的な表現が必要不可欠です。
このような観点から、ヒトの生物学、遺伝子診断・遺伝子治療を含むゲノム医学に即戦力として利用できるソフトウェアとしてMutationViewは開発されました。現バージョンの主な特徴は以下の通りです。
(1) 染色体イディオグラムを表示し、疾患遺伝子のマップを一覧表示します。それらのOMIM情報も表示できます。
(2) 身体・臓器の模式図上に、各臓器で起こる遺伝子/疾患名を配置し表示できます。
(3) (1)または(2)の方法で指定した遺伝子に関して、横軸を塩基数として構造を表示し、様々な突然変異の位置を表示します。横軸の拡大・縮小は任意で、塩基配列も表示できます。縦軸は症例数によるヒストグラム、変異のタイプによる分類、遺伝形質による分類等を選択して表示できます。
(4) 塩基配列を正常と変異で比較表示し、変異部位近傍の制限酵素認識配列の変化を明示します。
(5) 突然変異のデータ集積は疾患ごとに個別の研究室で行われているのが現状ですが、本システムを共用することによってインターネット経由でそれらを分散データベースとして相互利用できます。
2. システム動作環境
現在、MutationViewのJava versionに対応しているWWWブラウザは次の通りです。
|
OS |
WWWブラウザ |
|
Windows XP/Vista/7/8 + jre1.5.0_12-b4 or later
|
Internet Explorer 10 (without Metro UI (Windows 8)) |
|
Internet Explorer 9 |
|
|
Internet Explorer 8 |
|
|
Firefox 4.0.1 or later |
|
|
Google Chrome 12.0.742.100 or later |
|
|
Safari 5.0 or later |
|
|
Opera 11.11 or later |
|
|
MacOS X 10.4/5/6/7 + jre1.5.0_16 to 1.6.0_45-b06-451 |
Firefox 5.0 or later |
|
Safari 3.2.1 or later |
|
|
Google Chrome |
3. 操作マニュアル
このマニュアルはWindowsにおけるボタン操作を前提として記述しています。Macintoshをお使いの場合は下記の表を参考にしてマッキントッシュのボタン操作に置き換えてご利用ください。UNIXにおけるボタン操作はWindowsと同様です。
|
Windows |
左クリック |
右クリック |
[Ctrl]+クリック |
|
Macintosh |
クリック |
[Shift]+クリック |
[Ctrl]+クリック |
|
UNIX |
左クリック |
右クリック |
[Ctrl]+クリック |
4. スタート画面
スタート画面から検索方法を選択します。全染色体入力遺伝子一覧メニューから検索する場合は[Chromosome Overview]ボタンを、疾患原因遺伝子がマップされている染色体から検索する場合は[Chromosome Details]ボタンを、疾患発症部位から検索する場合は[Anatomy]ボタンを押します。
[OMIM]ボタンはOMIMに登録されている疾患遺伝子が検索でき、検索結果はOMIMページにリンクしています。

図1スタート画面
5. 目的遺伝子の検索入り口
5.1. Chromosome Overviewメニューからの検索
[Chromosome Overview]ボタンを押すと全染色体入力遺伝子一覧メニューが現れます。
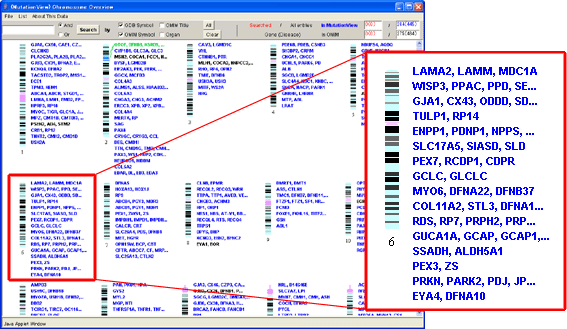
図2-1全染色体入力遺伝子一覧メニュー
全染色体入力遺伝子一覧メニューは、MutationViewに登録されている遺伝子の一覧です。1番からX、Y染色体のイディオグラムが表示され、イディオグラムの右側に、登録されている遺伝子の遺伝子記号が青色で表示されます。これから登録予定の遺伝子は黒色で表示されます。遺伝子記号の上にマウスポインタを合わせると、その遺伝子の染色体マップ領域がイディオグラムと直線で結ばれます。その状態で遺伝子記号をクリックすると遺伝子構造ウィンドウ(後述6.)が表示されます。遺伝子記号を右クリックすると遺伝子名詳細ウィンドウ(後述5.1.3.)が表示されます。イディオグラムそのものをクリックするとその染色体番号のイディオグラム遺伝子リスト(後述5.2.1)が表示されます。
画面右上の数字[In MutationView]は、MutationViewに登録されている遺伝子数を示し、カッコ内の数字はそれらの遺伝子を原因とする疾患数を示します。下段の数字はMutationViewに登録されている遺伝子数とこれから登録予定の遺伝子数を合計したものです。
[List]メニューで[Disease->OMIM Symbol]を選択すると全染色体入力遺伝子一覧メニューから全染色体入力疾患一覧メニューに変更されます。全染色体入力疾患一覧メニューではMutationViewに登録されている遺伝子を原因とする疾患を表示します。染色体イディオグラムの右側に登録されている疾患のOMIM記号が青色で表示されます。これから登録予定の疾患は黒色で表示されます。OMIM記号を右クリックすると疾患名詳細ウィンドウ(後述5.1.4)が表示されます。
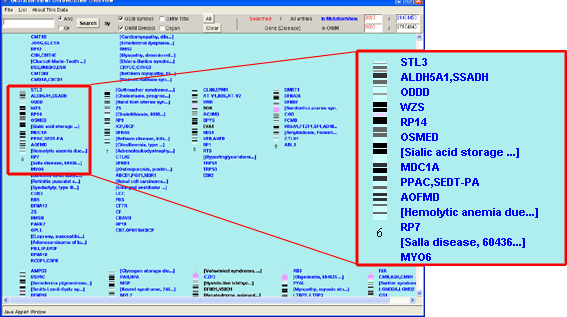
図2-2全染色体入力疾患一覧メニュー
5.1.1. 検索機能
ウィンドウ上部にある検索ツールバーを用いると遺伝子名、疾患名、発症部位によって全染色体入力遺伝子一覧メニューの中から目的とする遺伝子を検索することができます。
[1]検索ツールバーボタンの説明
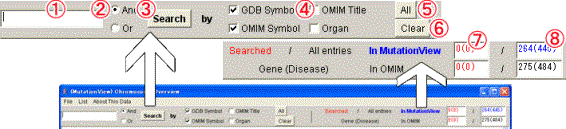
図2-3検索ツールバー
(1) 検索キーワード入力フィールド:検索キーワードを入力します。複数のキーワードを入力するときはスペースで区切ってキーワードを入力します。
(2) 検索モードラジオボタン:複数のキーワードを入力したとき、すべてのキーワードに一致させたい場合は[And]を選択し、キーワードのうちいずれかに一致させたい場合は[Or]を選択します。デフォルトでは[And]が選択されています。
(3) [Search]ボタン:検索を実行します。
(4) 検索フィールドチェックボックス:検索対象とする項目を指定します。デフォルトでは[GDB Symbol]と[OMIM Symbol]を対象としています。
(5) チェック[All]ボタン:(4)のチェックボックスをすべて選択します。
(6) チェック[Clear]ボタン:(1)のキーワードフィールドのキーワードを削除し、(4)検索対象ボックスの指定を解除します。検索結果が表示されている場合は検索結果の表示を解除します。
(7) 検索ヒット数表示フィールド:上段に検索条件に該当する遺伝子数を表示します。下段はこれからMutationViewに登録予定の遺伝子数を足したものです。カッコ内の数字はそれらの遺伝子を原因とする疾患数を示します。
(8) 登録遺伝子数表示フィールド:上段にMutationViewに登録されている遺伝子数の合計を表示します。下段はこれからMutationViewに登録予定遺伝子の数を足したものです。カッコ内の数字はそれらの遺伝子が原因となる疾患数を示します。
[2]検索手順
(1) [1]-(1)の検索キーワード入力フィールドにキーワードを入力します。[1]-(2)の検索モードラジオボタンを選択します。
(2) [1]-(4)の検索フィールドチェックボックスから検索対象項目を選択します。
(3) [1]-(3)の[Search]ボタンを押すと、[1]-(7)の検索ヒット数表示フィールドに該当する遺伝子数が示され、遺伝子一覧で該当する遺伝子が赤色で表示されます。
(4) [1]-(6)の[Clear]ボタンを押すとキーワードの入力、検索項目の選択、及び検索結果の表示が解除されます。
5.1.2. メニュー機能
メニューバーにある機能を示します。
·[File]メニュー
- [Exit]:全染色体入力遺伝子一覧メニューを閉じます。
- [Save]:2.1.1検索機能で検索した遺伝子のリストと、それらの遺伝子の遺伝子名詳細ウィンドウに表示される情報を保存します。
·[List]メニュー
- [Gene->OMIM Symbol]:MutationViewに登録されている遺伝子の一覧(図2-1全染色体入力遺伝子一覧メニュー)を表示します。デフォルトで選択されています。
- [Disease->OMIM Symbol]:MutationViewに登録されている遺伝子を原因とする疾患の一覧(図2-2全染色体入力疾患一覧メニュー)を表示します。
·[About This Data]メニュー「Curator」:OMIMについての情報を表示します。
5.1.3. 遺伝子名詳細ウィンドウ
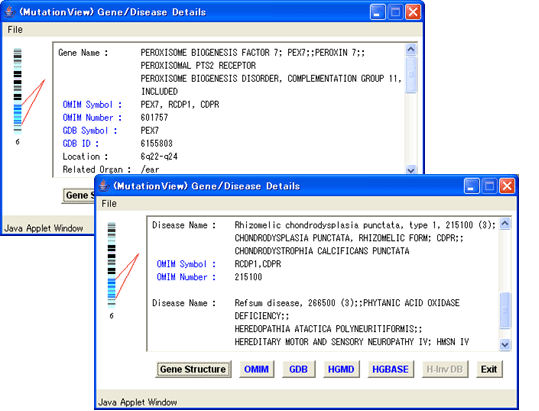
図2-4遺伝子名詳細ウィンドウ
遺伝子名詳細ウィンドウは遺伝子に関する情報を表示するウィンドウです。遺伝子名、遺伝子のOMIM記号、OMIM番号、GDB記号、GDB番号、染色体マップ領域、疾患発症器官等の遺伝子に関する情報と、この遺伝子を原因とする疾患の疾患名、OMIM記号及びOMIM番号を表示します。
ウィンドウ下部のボタンは遺伝子構造ウィンドウの表示や他のデータベースへのリンクボタンです。[OMIM]ボタンを押すと、NCBIにリンクし該当するOMIMエントリーを表示します。同様に[GDB]ボタンを押すとGDBにおける該当するエントリーにリンクし、[HGMD]ボタンを押すとHGMDおける該当するエントリーにリンクし、[HGBASE]ボタンを押すとHGBASEおける該当するエントリーにリンクします。[Gene Structure]ボタンを押すと遺伝子構造ウィンドウ(後述6.)を表示します。[Exit]ボタンを押すとこのウィンドウを閉じます。
5.1.4. 疾患名詳細ウィンドウ
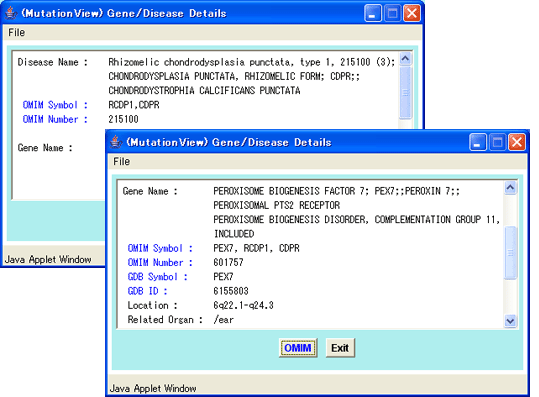
図2-5疾患名詳細ウィンドウ
疾患名詳細ウィンドウは疾患名、OMIM記号、OMIM番号と、疾患の原因となる遺伝子の遺伝子名、OMIM記号、OMIM番号、GDB記号、GDB番号、染色体マップ領域及び疾患発症器官を表示します。ウィンドウ下部[OMIM]ボタンを押すと、NCBIにリンクし該当するOMIMエントリーを表示します。[Exit]ボタンを押すとこのウィンドウを閉じます。
5.2. Chromosome Detailsメニューからの検索
[Chromosome Details]ボタンを押すと染色体選択メニューが表示されます。一つ一つの染色体の図がボタンになっており、目的とする疾患/遺伝子がマップされている染色体のボタンを押すと、その染色体の詳細を示したイディオグラム遺伝子リスト(後述5.2.1)が表示されます。[mitochondorion]ボタンを押すとミトコンドリアゲノムデータベースMITOMAPにリンクします。

図2-6染色体選択メニュー
5.2.1. イディオグラム遺伝子リスト
イディオグラム遺伝子リストはMckusickのデータをもとに作られ、選択されている染色体上に存在する単一遺伝子疾患原因遺伝子の遺伝子名の一覧を表示します。
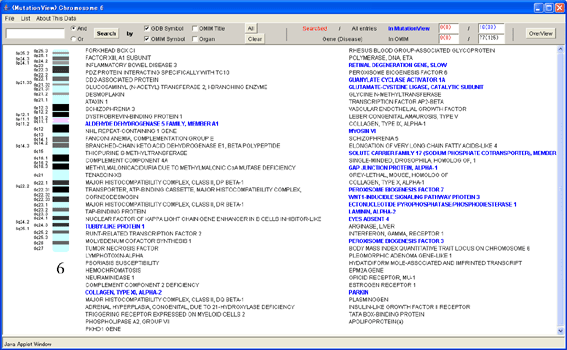
図2-7イディオグラム遺伝子リスト
マウスポインタを遺伝子名に合わせると、その遺伝子のマップ領域がイディオグラムと線で結ばれます。その状態で遺伝子名を右クリックすると遺伝子名詳細ウィンドウ(前述5.1.3)が表示されます。青色の文字で表示された遺伝子名/疾患名は現在MutationViewに登録されているものを表し、クリックすると遺伝子構造ウィンドウ(後述6.)が表示されます。右上の[Chromosome Overview]ボタンを押すと全染色体入力遺伝子一覧メニュー(前述5.1)が表示されます。
デフォルトでは遺伝子名が表示されていますが、[List]メニューで[Gene->OMIM Symbol]を選択すると、遺伝子名からOMIM記号に変更されます(後述5.2.3)。
[List]メニューで[Disease->OMIM Title]を選択すると、この染色体にマップされている単一遺伝子疾患の疾患名の一覧が表示されます(後述5.2.3)。疾患名を右クリックすると疾患名詳細ウィンドウ(前述5.1.4)が表示されます。疾患名は、[List]メニューで[Disease->OMIM Symbol]を選択すると疾患のOMIM記号に変更されます。
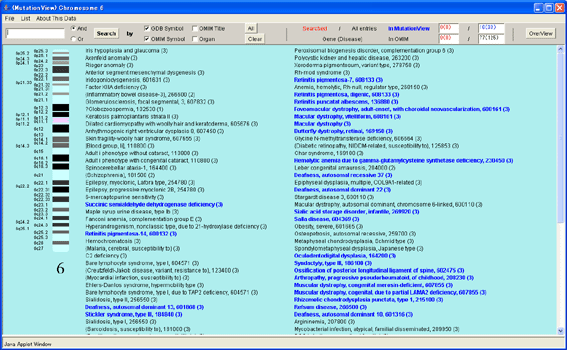
図2-8イディオグラム疾患リスト
5.2.2. 検索機能
ウィンドウ上部にある検索ツールバーを用いると遺伝子名、疾患名、発症部位によって遺伝子名/疾患名リストの中から目的とする遺伝子を検索することができます。
[1]検索ツールバーボタンの説明
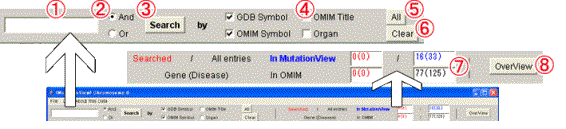
図2-9検索ツールバー
(1)〜(6)のボタンの機能は前述5.1.1と同様です。
(7) 検索数・登録数表示フィールド:上段の[In MutationView]は、この染色体でMutationViewに登録されている遺伝子数(青色の数字。カッコ内は疾患数。)とそのうち検索条件に該当する遺伝子数(赤色の数字。カッコ内は疾患数。)を表示します。下段の[In OMIM]はこの染色体でOMIMに登録されている疾患遺伝子数(黒色の数字。カッコ内は疾患数。)と、そのうち検索条件に該当する遺伝子数(赤色の数字。カッコ内は疾患数。)を表示します。
(8) [Overview]ボタン:全染色体入力遺伝子一覧メニュー(前述5.1)を表示します。
[2]検索手順
検索条件の入力方法は、5.1.1に記述した全染色体入力遺伝子一覧メニューにおける検索方法と同様です。[Search]ボタンを押すと、検索数・登録数表示フィールドにこの染色体で検索条件に該当する遺伝子数とそのうちMutationViewに登録されている遺伝子数が示されます。検索条件に該当する遺伝子は一覧中に赤色で表示されます。MutationViewに登録されているものは太字で、登録されていないものは細字で表示されます。
5.2.3. メニュー機能
メニューにある機能を以下に示します。
·[File]メニュー
- [Exit]:イディオグラム遺伝子リストを閉じます。
- [Save]:現在選択している染色体にマップする疾患遺伝子の一覧と、それらの遺伝子の遺伝子名詳細ウィンドウで表示される情報を保存することができます。
·[List]メニュー
- [Gene->OMIM Title]:この染色体に存在する単一遺伝子疾患原因遺伝子の一覧を遺伝子名で表示します(図2-7イディオグラム遺伝子リスト)。デフォルトで選択されています。
- [Gene->OMIM Symbol]:この染色体に存在する単一遺伝子疾患原因遺伝子の一覧をOMIM記号で表示します。
- [Disease->OMIM Title]:この染色体にマップされている単一遺伝子疾患の一覧を疾患名で表示します(図2-8イディオグラム疾患リスト)。
- [Disease->OMIM Symbol]:この染色体にマップされている単一遺伝子疾患の一覧をOMIM記号で表示します。
·[About This Data]メニュー「Curator」:OMIMについての情報を表示します。
5.3. Anatomyメニューからの検索
スタート画面で[Anatomy]ボタンを押すとAnatomyメニューが現れます。Anatomyメニューでは人体模式図に器官名、組織名のボタンを配置しており、疾患発症部位から疾患原因遺伝子や疾患の検索を行うことができます。
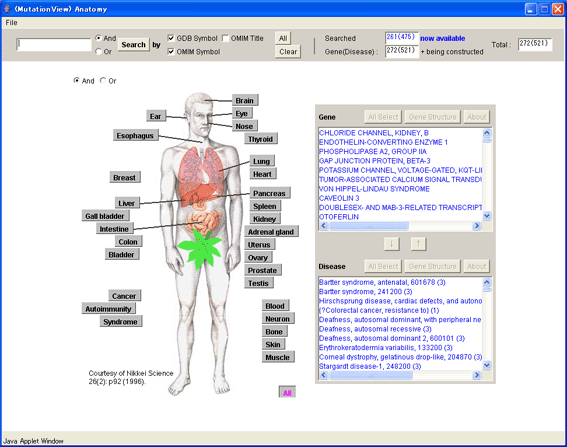
図2-10Anatomyメニュー
器官名、組織名のボタンを押すと、MutationViewに登録されている遺伝子の中で、その部位で発症する疾患の原因となる遺伝子が表示されます。表示された遺伝子名をクリックすると遺伝子構造ウィンドウ(後述6.)が表示されます。右クリックすると遺伝子名詳細ウィンドウ(前述5.1.3)が表示されます。
5.3.1. 検索機能
器官名、組織名のボタンから疾患発症部位から疾患原因遺伝子や疾患の検索を行うことができ、さらにキーワード入力によって結果の絞込みを行うことがきます。
[1]ボタンの説明
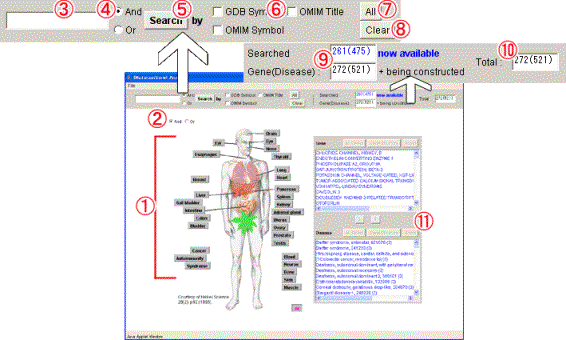
図2-11 Anatomyメニュー検索機能
(1) 器官名・組織名ボタン:器官名、組織名がボタンになっており、目的とする器官や組織を選択できます。デフォルトでは[All]ボタンが選択されておりすべての器官・組織を対象としています。 [Ctrl]と同時に器官名・組織名ボタンをクリックすることで複数の器官、組織を選択することが可能です。
(2) 器官名・組織名検索モードラジオボタン:複数の器官名・組織名ボタンを選択したとき、すべての器官、組織を対象とする場合は[And]を選択し、器官、組織のうちいずれかを対象とする場合は[Or]を選択します。デフォルトでは[And]が選択されています。
(3) 検索キーワード入力フィールド:検索キーワードを入力します。複数のキーワードを入力するときはスペースで区切ってキーワードを入力します。
(4) 検索モードラジオボタン:複数のキーワードを入力したとき、すべてのキーワードに一致させたい場合は[And]を選択し、キーワードのうちいずれかに一致させたい場合は[Or]を選択します。デフォルトでは[And]が選択されています。
(5) [Search]ボタン:キーワード検索を実行します。
(6) 検索フィールドチェックボックス:検索対象とする項目を指定します。デフォルトでは[GDB Symbol]と[OMIM Symbol]を対象としています。
(7) チェック[All]ボタン:(6)のチェックボックスをすべて選択します。
(8) チェック[Clear]ボタン:(3)のキーワードフィールドのキーワードを削除し、(6)検索対象ボックスの指定を解除します。検索結果が表示されている場合は検索結果の表示を解除します。
(9) 検索ヒット数表示フィールド:上段に検索条件に該当する遺伝子数を表示します。下段はこれからMutationViewに登録予定遺伝子の数を足したものです。カッコ内の数字はそれらの遺伝子が原因となる疾患の数を示します。
(10) 登録遺伝子数表示フィールド: MutationViewに登録されている遺伝子数の合計を表示します。カッコ内の数字はそれらの遺伝子が原因となる疾患の数を示します。
(11) 検索結果表示パネル:検索条件を満たす遺伝子の遺伝子名と、その遺伝子を原因とする疾患名を表示します(後述5.3.1 [3]で詳細を記述)。
[2]検索手順
(1) 目的とする器官や組織を?@の器官名・組織名ボタンから選択します。複数のボタンを選択した場合は[1]-(2)の器官名・組織名検索モードラジオボタンを選択します。
(2) [1]-(9)の検索ヒット数表示フィールドに該当する遺伝子数が表示され、[1]-(11)の検索結果表示パネル(後述5.3.1 [3])に該当する遺伝子名、疾患名が表示されます。
(3) キーワードによりさらに絞り込んだ検索を行うには、[1]-(3)の検索キーワード入力フィールドにキーワードを入力します。
(4) [1]-(4)の検索モードラジオボタンを選択します。
(5) [1]-(6)の検索フィールドチェックボックスから検索対象項目を選択します。
(6) [1]-(5)の[Search]ボタンを押すと、[1]-(9)の検索ヒット数表示フィールドに該当する遺伝子数が表示され、[1]-(11)の検索結果表示パネル(後述5.3.1 [3])に該当する遺伝子名、疾患名が表示されます。
(7) [1]-(11)の検索結果表示パネルから遺伝子や疾患を選択し遺伝子構造ウィンドウを表示します。
(8) キーワード検索を解除するには[1]-(3)の検索キーワード入力フィールドをクリアし[1]-(5)の[Search]ボタンを押します。
[3]検索結果表示パネルの説明
検索結果表示パネルは検索条件を満たす遺伝子の遺伝子名と、その遺伝子を原因とする疾患名を表示します。遺伝子名と疾患名は相互に結びついており、遺伝子名から対応する疾患名の表示、疾患名から対応する遺伝子名の表示を行うことができます。遺伝子名、疾患名を選択してから遺伝子構造ウィンドウを表示します。

図2-12検索結果表示パネル
(1) 遺伝子名リスト:検索条件を満たす遺伝子の遺伝子名を表示します。青色の遺伝子名はMutationViewに登録されている遺伝子で、黒色の遺伝子名は登録予定の遺伝子です。遺伝子名を右クリックすると遺伝子名詳細ウィンドウ(前述5.1.3)が表示されます。
(2) 疾患名リスト:検索条件を満たす遺伝子を原因とする疾患の疾患名を表示します。青色の疾患名は原因遺伝子がMutationViewに登録されている疾患で、黒色の遺伝子名は登録予定の疾患です。疾患名を右クリックすると疾患名詳細ウィンドウ(前述5.1.4)が表示されます。
(3) [↓]ボタン:(1)の遺伝子名リストで選択した遺伝子に対応する疾患を(2)の疾患名リストに絞り込んで表示します。
(4) [↑]ボタン:(2)の疾患名リストで選択した疾患に対応する遺伝子を(1)の遺伝子名リストに絞り込んで表示します。
(5) [All Select]ボタン:(4)のボタンによる疾患対応遺伝子の絞込みを解除し、検索条件を満たす遺伝子をすべて表示します。
(6) [GeneStructureWindow]ボタン:(1)の遺伝子名リストの中で選択された遺伝子の遺伝子構造ウィンドウを表示します。
(7) [About]ボタン:(1)の遺伝子名リストの中で選択された遺伝子の遺伝子名詳細ウィンドウ(前述5.1.3)を表示します。
(8) [All Select]ボタン:(3)のボタンによる遺伝子対応疾患の絞込みを解除し、検索条件を満たす疾患をすべて表示します。
(9) [GeneStructureWindow]ボタン:(2)の疾患名リストの中で選択された疾患の原因遺伝子の遺伝子構造ウィンドウを表示します。
(10) [About]ボタン:(2)の疾患名リストの中で選択された疾患の疾患名詳細ウィンドウ(前述5.1.4)を表示します。
6. 遺伝子構造ウィンドウ
遺伝子構造ウィンドウは、各遺伝子の変異情報をグラフィカルに表示するMutationViewの中核的ウィンドウです。
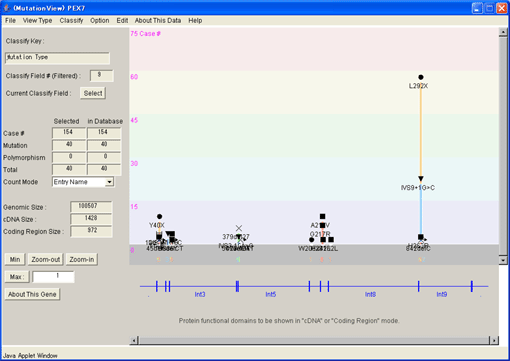
図3-1遺伝子構造ウィンドウ
6.1. ウィンドウの基本構造
ウィンドウの横軸は塩基配列数を長さの単位とし、デフォルトでは遺伝子構造を示します。横軸の表示はメニュー[View Type]からcDNA 、翻訳領域に変更することもできます(後述6.3メニューバー機能)。スクロールバーを左右に移動すると表示する範囲を変更することができます。ウィンドウを拡大すると横軸の表示範囲が拡大され、最大拡大で塩基配列が表示されます(拡大、縮小方法は後述6.2.1)。このときエキソンの塩基配列は大文字(A,T,G,C)で表示され、さらに翻訳領域ではコドンに対応するアミノ酸が並べて表示されます。イントロンの塩基配列は小文字(a,t,g,c)で表示されます。
縦軸は横軸の塩基位置における変異の症例数を示し、ヒストグラムによってビジュアル的に表示します。縦軸の単位はメニュー[View Type]から発症率などに変更することもできます(後述6.3メニューバー機能)。
ヒストグラムはクラシファイで設定された分類により色分けされています(クラシファイについては後述6.4)。色分けの詳細はツールバーの[Current Classify Field]の[Select]ボタンを押すと表示されます。ヒストグラムの先端にある●、■などの記号は変異の種別を表しています。[Help]メニュー[Symbol Table]を選択すると記号と変異種別の一覧が表示されます。
6.2. ウィンドウ基本操作方法
6.2.1. 拡大・縮小機能
以下に示す方法によって横軸の表示スケールを変更することができます。
a) ポインタにより拡大・縮小する位置を選択する・・・拡大したいエキソン、イントロンの位置にマウスポインタを移動し、そこで右クリックするとマウスポインタの位置を基準として一段階拡大されます。右クリックするたびに一段階拡大され、塩基配列が表示されたところが最大拡大です。[Ctrl]+クリックで一段階ずつ縮小されます。
b) 拡大・縮小ボタンを用いる・・・ウィンドウ左のツールバーにある[Zoom-in]ボタンを押すとウィンドウの中央を基準として一段階拡大されます。[Zoom-out]ボタンを押すとウィンドウの中央を基準として一段階縮小されます。
c) 最大・最小ボタンを用いる・・・最大拡大したい場所にマウスポインタを合わせクリックします。するとツールバーの[Max]ボタンの隣にマウスポインタが示す位置の塩基番号が表示されます。塩基番号が指定された状態で[Max]ボタンを押すと、その位置を中心として最大拡大されます。[Min]ボタンを押すと最大縮小されます。
6.2.2. 変異詳細ウィンドウの表示
変異種別記号をクリックすると変異詳細ウィンドウが現れ、変異の詳細情報が表示されます。ウィンドウの説明を以下に示します。
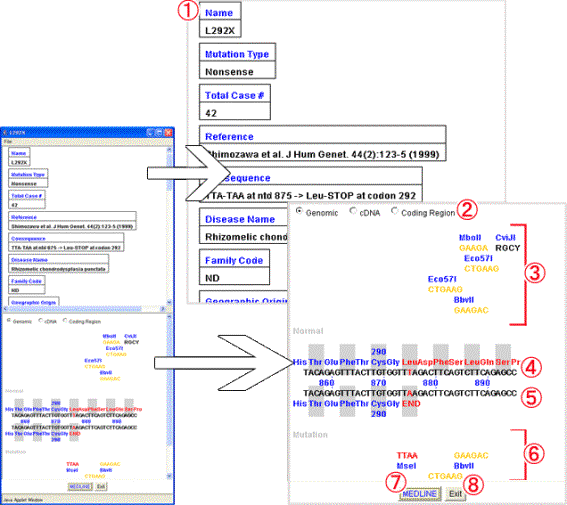
図3-2(a) 変異詳細ウィンドウ
(1) 変異詳細表示フィールド:変異名、文献、変異による結果及び変異種別を表示します。
(2) 表示モードラジオボタン:(4)、(5)の変異前後の塩基配列・アミノ酸配列を、 [Genomic]、[cDNA]、[Coding Region]のボタンに応じて、ゲノム構造、cDNAまたは翻訳領域に変更します。デフォルトでは[Genomic]が選択されています。
(3) 変異前制限酵素サイト:制限酵素サイトと制限酵素名を表示します。変異後と異なる制限酵素サイトを赤色で表示します。
(4) 変異前塩基・アミノ酸配列:変異前の塩基配列と対応するアミノ酸配列を表示し、変異部分は赤色で表示します。
(5) 変異後塩基・アミノ酸配列:変異後の塩基配列と対応するアミノ酸配列を表示し、変異部分は赤色で表示します。
(6) 変異後制限酵素サイト:制限酵素サイトと制限酵素名を表示します。
(7) [Exit]ボタン:このウィンドウを閉じます。
(8) [MEDLINE]ボタン:この変異の引用文献のMEDLIN要旨にリンクします。
また、スプライシング変異のなかで、ドナーサイトもしくはアクセプターサイト近傍の変異にについては、変異の結果予測される構造が表示されます。
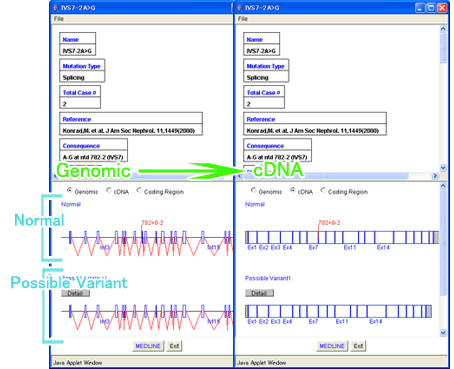
図3-2(b) スプライシングの変異詳細ウィンドウ
6.2.3. 塩基番号の表示
マウスポインタをウィンドウ上で移動するとマウスポインタのある場所の塩基番号が表示されます。クリックするとウィンドウが開き、その位置のエキソンまたはイントロンに含まれる塩基数が表示されます。[OK]ボタンを押すとこのウィンドウを閉じます。塩基番号を非表示にする場合は[Option]メニューの[Base#]のチェックをはずします。
6.3. メニューバー機能
メニューバーにある機能を以下に示します。
·[File]メニュー
- [Exit]:遺伝子構造ウィンドウを閉じます。
·[View Type]メニュー
- [Genomic]、[cDNA]、[Coding Region]を選択すると横軸の表示を変更することができます。
[Case #]、[Zone plot]、[Calculated Frequency]、[Literature Frequency]は縦軸の表示方法を変更します。
- [Case #]:変異ごとの症例数をヒストグラムで表示します。
- [Zone plot]:クラシファイで指定された分類中の種別ごとにゾーンを区切り、種別に対応したゾーンに変異を点で表示します。
- [Calculated Frequency]:この遺伝子の変異の総数に対するそれぞれの位置における変異の頻度をヒストグラムで表示します。
- [Literature Frequency]:論文に記載されている頻度をヒストグラムで表示します。
- [Count by Mutation Name]:変異名が同一のものを一つの変異として表示します。デフォルトではチェックされています。変異名が同一であっても詳細の異なる変異を別々に表示させたいときはチェックをはずします。
- [Probe]:横軸の遺伝子構造に対応したプローブ記号を表示します。プローブ記号をクリックすると、プライマーの塩基配列、反応条件、引用文献などの情報を表示したウィンドウが現れます。
·[Classify]:クラシファイウィンドウを表示します。突然変異種別や人種、発症年齢などの条件で表示を絞り込むことができます(詳細は後述6.4)。
·[Option]メニュー
- [Search]:塩基配列検索ウィンドウを表示します(詳細は後述6.5)。
- [Base #]:マウスポインタをウィンドウ上で移動させたときにポインタのある場所の塩基番号を表示します。デフォルトでは選択されています。
- [Point Mutation]:ポイントミューテーションの存在する塩基位置に縦線を表示します。
·[About This Data]メニュー
- [Curator]:データの由来を表示します。
- [Sequence Data]:この遺伝子の塩基配列の由来を表示します。
·[Help]メニュー[Symbol Table]:変異の記号の意味を表示します。
6.4. クラシファイ機能
[Classify]メニューを選択するとクラシファイウィンドウが表示されます。クラシファイ機能を使用すると表示データの条件選択や分類表示を行うことができます。
[1]クラシファイウィンドウのボタンの説明
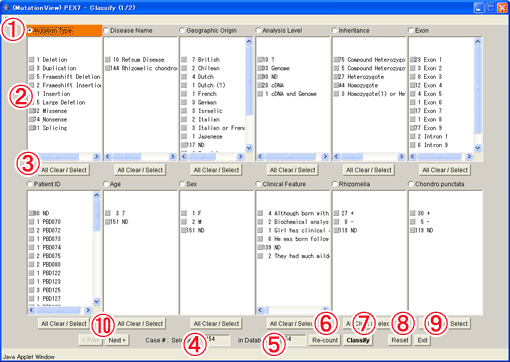
図3-3クラシファイウィンドウ
(1) 分類項目ボタン:選択された分類によって遺伝子構造ウィンドウが表示されます。またツールバーの[Classify Key]が変更されます。デフォルトでは[Mutation Type]が選択されています。
(2) 分類種別リスト:各分類における種別の一覧が表示されます。クリックすることで遺伝子構造ウィンドウに表示したい種別を選択、選択解除することができます。デフォルトでは”polymorphism”を除いた変異が選択されています。
(3) [All Clear/ Select]ボタン:分類項目における種別をすべて選択解除またはすべて選択します。
(4) 選択変異数:選択された条件における変異数を表示します。
(5) 変異合計異数:登録されている変異数を表示します。
(6) [Re-count]ボタン:指定した条件で変異数を再計算します。
(7) [Classify]ボタン:指定した条件で遺伝子構造ウィンドウを再表示します。
(8) [Reset]ボタン:選択した条件を解除します。
(9) [Exit]ボタン:このウィンドウを閉じます。
(10) [< Prev]ボタン/[Next >]ボタン:クラシファイ項目が複数のページに渡る場合にページを変更します。
[2]クラシファイの使用方法
(1) [1]-(1)の分類項目ボタンから縦軸に表示したい分類を選択します。
(2) [1]-(2)の分類種別リストから遺伝子構造ウィンドウに表示させたい種別を選択します。グループ化機能を用いると表示されている分類種別をグループ化することができます。グループ化したい種別を右クリックするとその種別が赤字になります。その状態で[Ctrl]+クリックするとグループ化ウィンドウが現れるのでグループ名を入力します。するとその分類中に入力した名称のグループが新たにできます。グループを解除するときは[Ctrl]+クリックでグループ化ウィンドウを表示し[Release]ボタンを押します。
(3) 分類、種別を選択した後に[Re-count]ボタンを押すと、選択された条件での変異数を再計算します。
(4) [Classify]ボタンを押すと、選択された条件に属する変異が遺伝子構造ウィンドウに表示されます。
6.5. 塩基配列検索機能
[Option]メニュー[Search]で塩基配列検索ウィンドウが表示されます。現在表示されている遺伝子構造の範囲で、入力した塩基配列の位置を検索することができます。
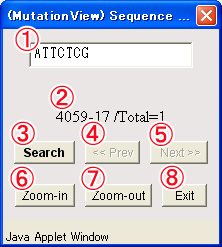
図3-4塩基配列検索ウィンドウ
[1]ボタンの説明
(1) 検索配列入力フィールド:検索したい配列を入力します。
(2) 検索結果表示フィールド:検索配列と一致する位置の合計数との塩基番号を表示します。一致する位置が複数ある場合は、最も5’末端に近い位置の塩基番号を表示します。
(3) [ Search]ボタン:検索を実行します。
(4) [<< Prev]ボタン:検索結果を5’末端側に移動します。
(5) [Next >>]ボタン:検索結果を3’末端側に移動します。
(6) [Zoon-in]ボタン:クリックすると検索結果の塩基番号を基準として遺伝子構造画面を一段階ずつ拡大します。
(7) [Zoom-out]ボタン:遺伝子遺伝子構造画面を一段階縮小します。
(8) [Exit]ボタン:このウィンドウを閉じます。
[2]検索手順
(1) 遺伝子構造ウィンドウで検索する対象の範囲を表示します。
(2) [1]-(1)の検索配列入力フィールドに塩基配列を入力します。
(3) [1]-(3)の[Search]ボタンを押します。
(4) [1]-(2)の検索結果表示フィールドに該当する位置が表示されます。この位置は遺伝子構造において縦棒でマークされます。
(5) 検索配列に一致する位置が複数ある場合は5’末端側から示され、[1]-(5)の[Next >>]ボタンで3’末端側に移動します。[1]-(4)の[<< Prev]ボタンで5’末端側に移動します。
(6) [1]-(6)の[Zoon-in]ボタンを押すと検索位置を基準として一段階ずつ拡大します。[1]-(7)の[Zoom-out]ボタンで一段階縮小します。
(7) [Exit]ボタンでこのウィンドウを閉じます。
6.6. ウィンドウツールバーの機能
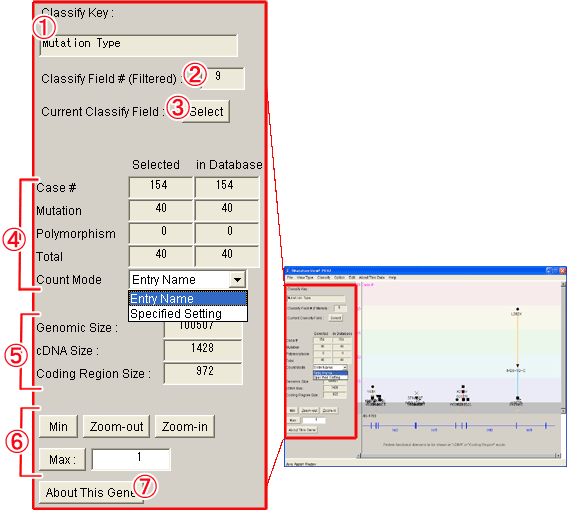
図3-5遺伝子構造ウィンドウツールバー
(1) [Classify Key]:クラシファイ(前述6.4)で設定されている分類の名称を表示します。
(2) [Classify Field]:クラシファイで設定されている分類のうち表示するように選択されている種別の数を表示します。
(3) [Select]ボタン:クラシファイで設定されている分類に属する種別の一覧が表示されます。表示させたい種別を選択すると、選択された種別の変異が遺伝子構造ウィンドウで表示されます。
(4) 変異数表示フィールド:以下の項目について、[Selected]ではクラシファイで設定されている条件における数を、[in Database]ではMutationViewに登録されている数を表示します。
·[Case#]:変異症例数の合計を示します。
·[Mutation]:変異数の合計を示します。
·[Polymorphism]:polymorphismの合計を示します。
·[Total]:変異数とpolymorphismの合計を示します。
·[Count Mode]:デフォルトでは[Entry Name]が選択され、同一変異名の変異を1つの変異として数えます。[Specified Setting]を選択すると同一変異名であっても詳細が異なるものは別の変異として数えます。
(5) 配列サイズ表示フィールド:遺伝子全体の、cDNA、翻訳領域の塩基数を表示します。
(6) 拡大・縮小ボタン
·[Zoom-out]ボタン:ウィンドウの中心を基準として一段階縮小します。
·[Zoom-in]ボタン:ウィンドウの中心を基準として一段階拡大します。
·[Min]ボタン:ウィンドウ画面を最小縮小します。
·[Max]ボタン:[Max]位置指定フィールドの位置を基準としてウィンドウを最大拡大します。
·[Max]位置指定フィールド:クリックした位置の塩基番号がこのフィールドに表示されます。
(7) [About This Gene]ボタン:遺伝子名詳細ウィンドウ(前述5.1.3)を表示します。
※このマニュアルにおける “クリック”はウィンドウズにおける左ボタンを意味し、マッキントッシュでは単なるクリックを意味します。“右クリック”はウィンドウズでは右ボタンを意味し、マッキントッシュでは[Shift]+クリックを意味します。
7. 実際の使用例1−目的遺伝子を検索する
スタート画面から、全染色体入力遺伝子一覧メニューから検索する場合は[Chromosome Overview]ボタンを、疾患原因遺伝子がマップされている染色体から検索する場合は[Chromosome Details]ボタンを、疾患発症部位から検索する場合は[Anatomy]ボタンを押します。
7.1. Chromosome Overviewメニューから遺伝子を検索する
7.1.1. 全染色体入力遺伝子一覧メニューの表示
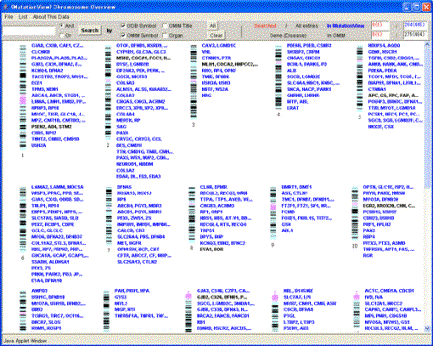
スタート画面で[Chromosome Overview]ボタンを押すと全染色体入力遺伝子一覧メニューが表示されます。
7.1.2. キーワード入力
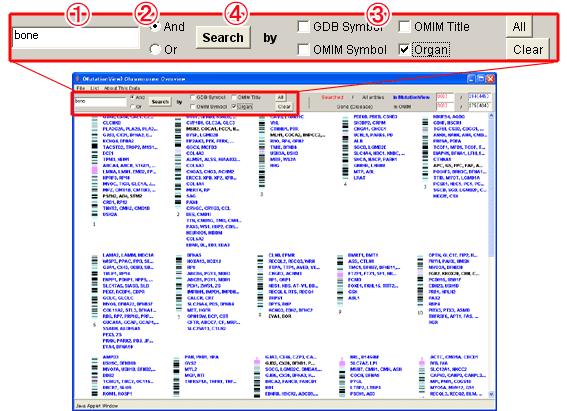
(1) 検索ツールバーに検索キーワード”bone”を入力します。
(2) (キーワードが複数の場合は[And]または[Or]を選択します。)
(3) 検索対象[Organ]を選択します。
(4) [Search]ボタンを押し検索を実行します。
7.1.3. 検索結果の表示
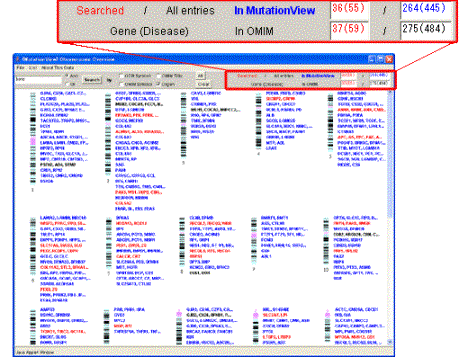
該当遺伝子が赤色で表示され、その合計数がツールバーに表示されます。
7.1.4. 検索結果からの選択
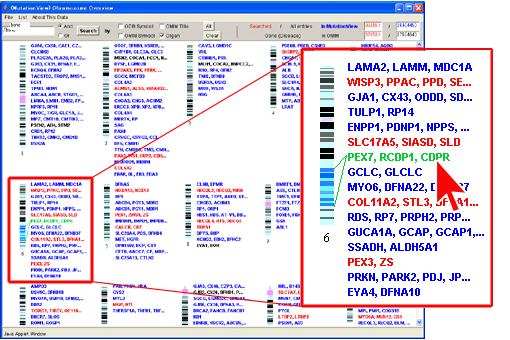
キーワードに該当した遺伝子の中から目的の遺伝子”PEX7”にマウスポインタを合わせます。すると、PEX7の遺伝子記号と6番染色体のイディオグラムのマップ領域が線で結ばれます。この状態で右クリックします。
7.1.5. 遺伝子名詳細ウィンドウの表示
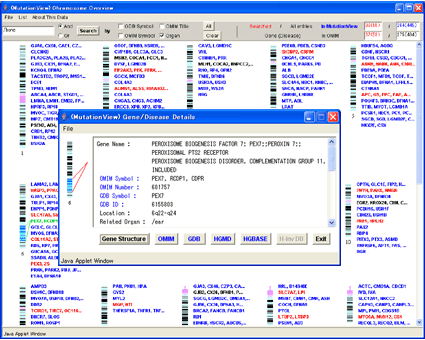
PEX7の遺伝子名詳細ウィンドウが表示されます。
7.2. Chromosome Detailsメニューから遺伝子を検索する
7.2.1. 染色体選択メニューの表示

スタート画面で[Chromosome Details]ボタンを押すと染色体選択メニューが表示されます。目的とする遺伝子/疾患がマップされている染色体のボタンを押します。ここでは6番染色体のボタンを押します。
7.2.2. イディオグラム遺伝子リストの表示
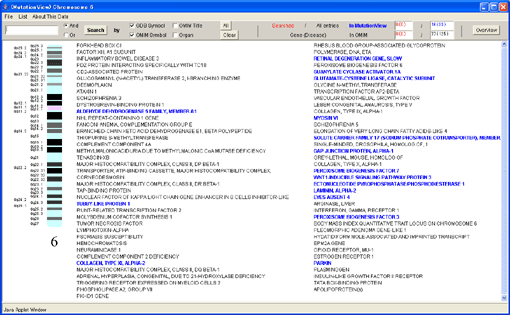
6番染色体のイディオグラム遺伝子リストが表示されました。
7.2.3. イディオグラム遺伝子リストからの検索
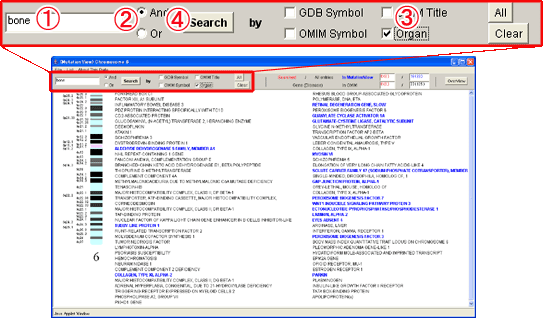
検索ツールバーを用いて6番染色体上の遺伝子から目的とする遺伝子をキーワードから検索します。
(1) 検索ツールバーに検索キーワード”bone”を入力します。
(2) (キーワードが複数の場合は[And]か[Or]を選択します。)
(3) 検索対象[Organ]を選択します。
(4) [Search]ボタンを押し検索を実行します。
7.2.4. 検索結果の表示
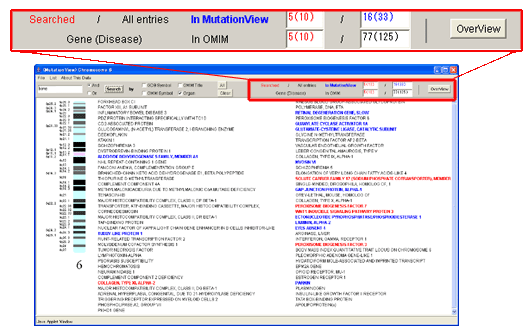
発症器官で “bone”のキーワードに該当する遺伝子数が赤字で表示されました。遺伝子一覧で該当遺伝子名が赤字で表示されました。
7.2.5. 検索結果からの選択
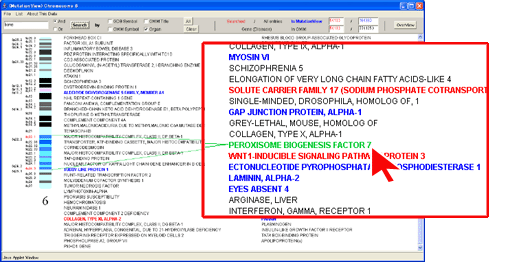
キーワードに該当した遺伝子の中から目的の遺伝子”PEROXISOME BIOGENESIS FACTOR 7”にマウスカーソルを合わせクリックします。すると遺伝子構造ウィンドウが表示されます。
7.2.6. イディオグラム疾患リストへの変更
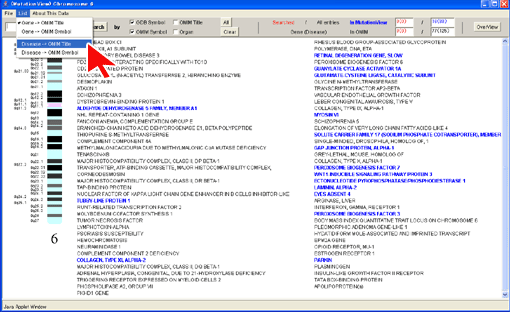
[List]メニューの[Disease-> OMIM Title]を選択します。
7.2.7. イディオグラム疾患リストの表示
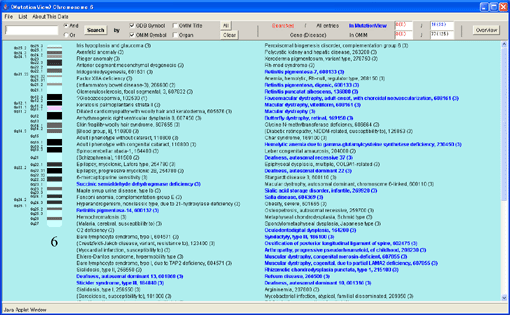
イディオグラム疾患リストに表示が変更されました。
7.2.8. イディオグラム疾患リストからの検索
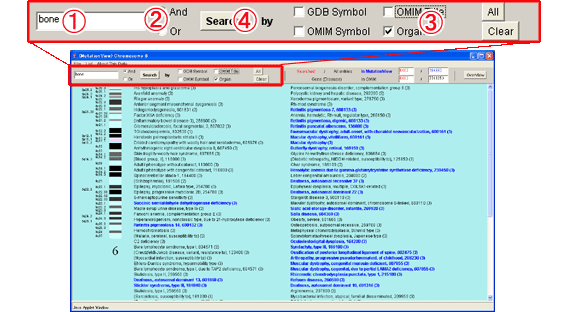
検索ツールバーを用いて6番染色体にマップされる疾患の中からキーワードによって目的とする疾患を検索します。
(1) 検索ツールバーに検索キーワード”bone”を入力します。
(2) (キーワードが複数の場合は[And]か[Or]を選択します。)
(3) 検索対象[Organ]を選択します。
(4) [Search]ボタンを押し検索を実行します。
7.2.9. 検索結果の表示
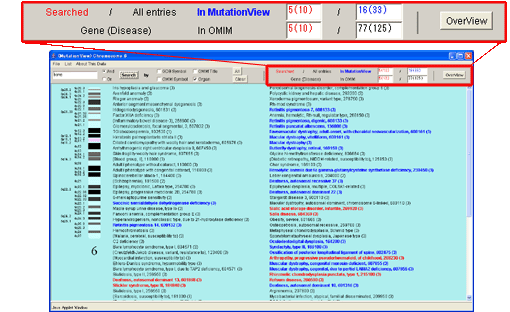
発症器官が“bone”のキーワードに該当する遺伝子数が赤字で表示されました。疾患一覧で該当疾患名が赤字で表示されました。
7.2.10. 検索結果からの選択
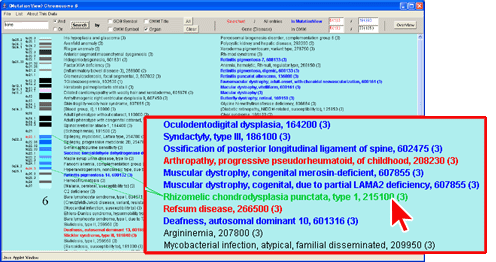
キーワードに該当した疾患の中から目的の疾患”Rhizomelic chondrodysplasia・・・”にマウスカーソルを合わせクリックします。すると遺伝子構造ウィンドウが表示されます。
7.3. Anatomyメニューから遺伝子を検索する
7.3.1. Anatomyメニューの表示
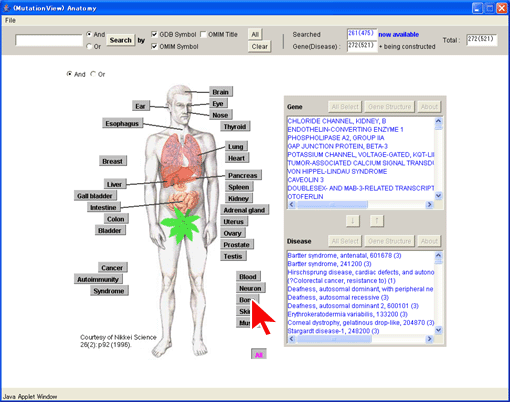
スタート画面で[Anatomy]ボタンを押すとAnatomyメニューが表示されます。目的とする疾患の発症部位のボタンを押します。ここでは[Bone]ボタンを押します。
7.3.2. 発症部位による検索結果
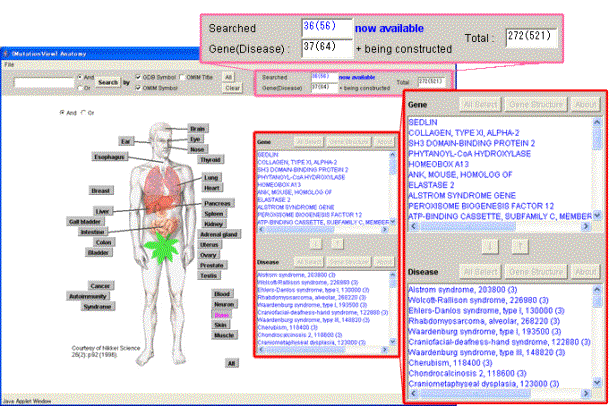
検索結果表示パネルに骨系統で発症する疾患名と遺伝子名が表示されました。ウィンドウ上部の検索ヒット数表示フィールドに遺伝子数と疾患数が表示されました。
7.3.3. キーワードによる絞込み検索
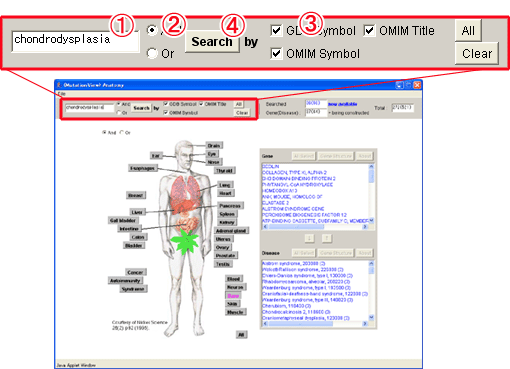
(1) 検索キーワード“chondrodysplasia”を入力します。
(2) (キーワードが複数の場合は[And]か[Or]を選択します。)
(3) 検索対象[OMIM Title]を選択します。
(4) [Search]ボタンを押し検索を実行します。
7.3.4. キーワードによる絞込み結果の表示
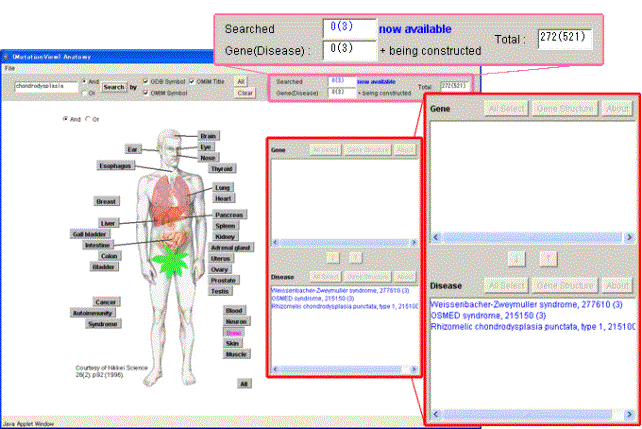
検索結果表示パネルに、骨系統で発症する疾患、遺伝子の中でキーワードを満たすものが表示されます。ウィンドウ上部の検索ヒット数表示フィールドに遺伝子数と疾患数が表示されました。
7.3.5. 疾患からの原因遺伝子の検索
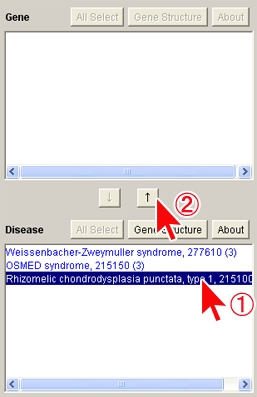
(1) 疾患名リストから目的とする疾患名を選択します。
(2) [↑]ボタンを押します。
7.3.6. 疾患から原因遺伝子の表示
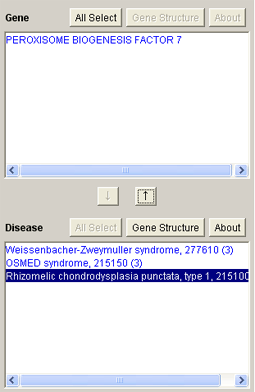
“Rhizomelic chondrodysplasia punctata, type1”の原因となる遺伝子が遺伝子名リストに表示されました。
7.3.7. 遺伝子の選択
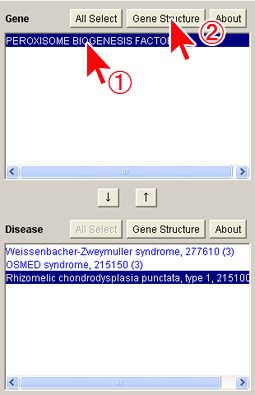
(1) 遺伝子名リストの“PEROXISOME BIOGENESIS FACTOR 7”をクリックします。
(2) [Gene Structure]ボタンを押すと遺伝子構造ウィンドウが表示されます。
8. 実際の使用例2−目的の遺伝子の変異情報を表示する

全染色体入力遺伝子一覧メニュー、イディオグラム遺伝子リスト、Anatomyメニューから目的とする遺伝子を選択すると、その遺伝子の変異情報を表示する遺伝子構造ウィンドウが現れます。ここでは6番染色体の遺伝子PEX7の遺伝子構造ウィンドウを表示しています。遺伝子構造ウィンドウの中ではクラシファイ機能を用いることで表示する変異情報の分類の変更、条件設定などができます。
8.1. クラシファイ機能の利用1−変異の付加情報から変異を分類表示する
デフォルトの遺伝子構造ウィンドウでは変異種別によって変異の分類が行われています。クラシファイ機能によって分類項目を変更し、疾患によって変異情報を表示する例を示します。
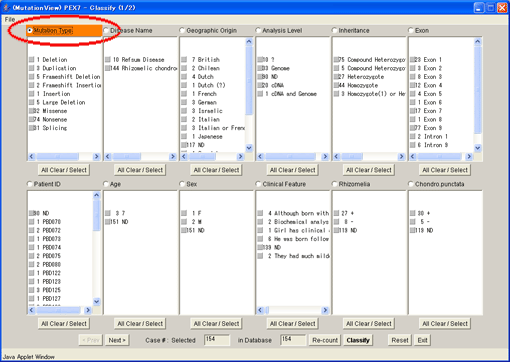
8.1.1. クラシファイウィンドウの表示
遺伝子構造ウィンドウで[Classify]メニューを選択しクラシファイウィンドウを表示させました。デフォルトで分類項目は”Mutation Type”が選択されています。
8.1.2. 分類項目の変更
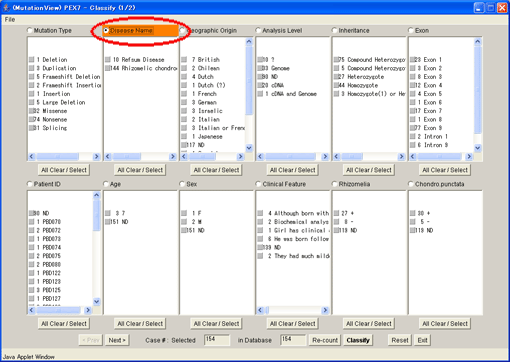
PEX7には“Disease Name”という分類項目があります。分類項目を“Disease Name”に変更し疾患に着目して変異を表示させることにします。[Classify]ボタンを押して変更した分類で遺伝子構造ウィンドウを表示させます。
8.1.3. 変更した分類項目での遺伝子構造ウィンドウの再表示
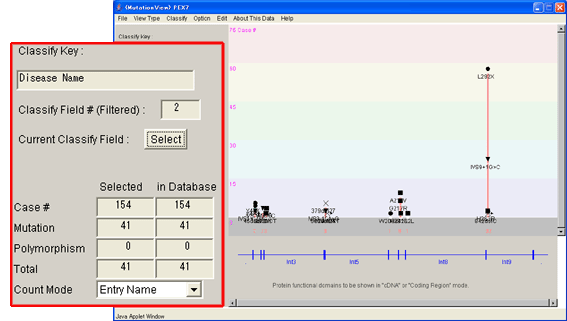
縦軸の表示が“Disease Name”に変更されました。[Classify Key]が“Disease Name”に変更され、変異数表示フィールドの値が更新されました。
8.1.4. 設定された分類項目の種別一覧の表示
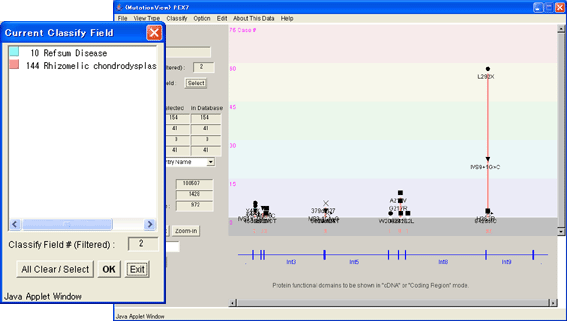
ツールバーの[Current Classify Field]の[Select]ボタンを押して、現在設定されている分類項目に属する種別を表示させました。種別の色分けはヒストグラムの色に対応しています。
8.1.5. 種別の絞込み表示
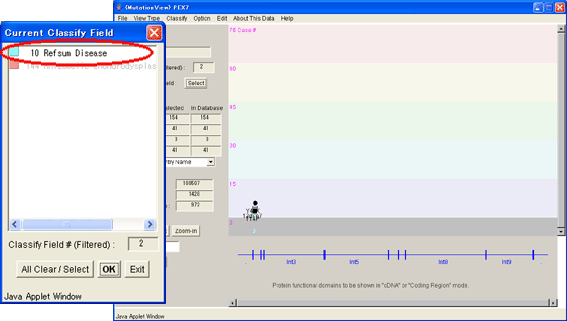
種別一覧で表示させたい種別を選択することができます。“Refsum Disease”を選択し、[OK]ボタンを押して“Refsum Disease”を発症する変異を表示させました。
8.1.6. 変異記号の表示
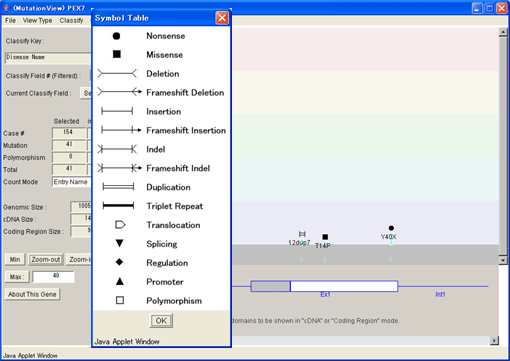
[Help]メニューを選択すると変異記号の説明が表示されます。
8.1.7. 遺伝子構造ウィンドウの拡大
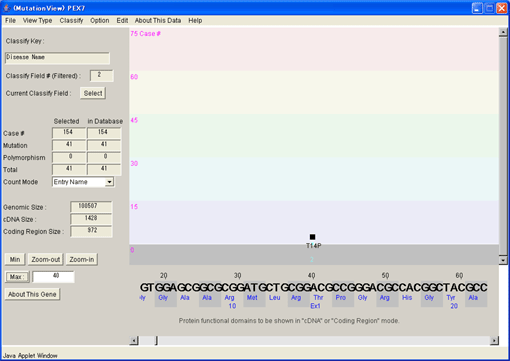
右クリックまたは、ツールバーの拡大ボタンでエキソン1を拡大し、最大拡大しました。塩基配列が表示され、その下にはコドンに対応するアミノ酸が表示されています。
8.1.8. 変異詳細ウィンドウの表示
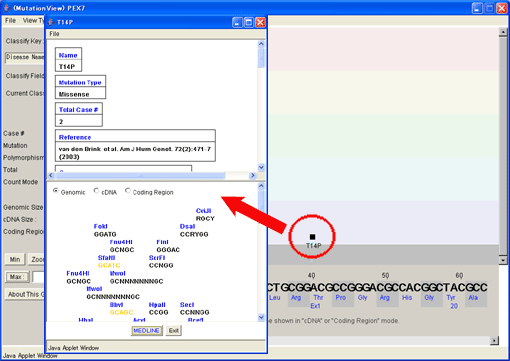
変異“T14P”の変異記号をクリックし変異詳細ウィンドウを表示させました。
8.1.9. プローブの表示
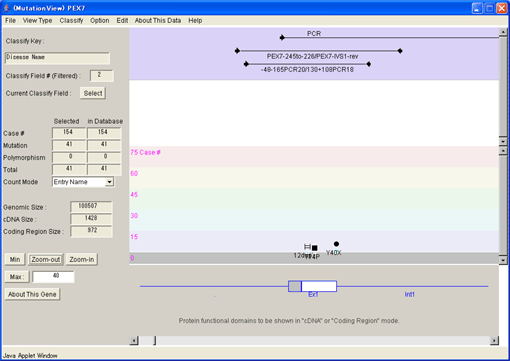
[View Type]メニューで[Probe]を選択しプローブの位置を表示させました。
8.1.10. プローブ詳細情報の表示
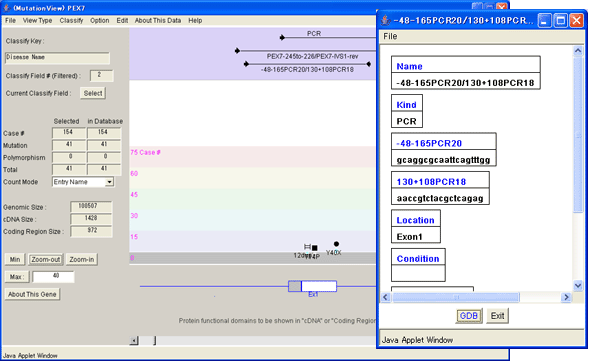
エキソン1を挟むプローブ“-48-165PCR20/130+108PCR18”をクリックし、プローブの詳細情報を表示させました。
8.1.11. 変異詳細詳細ウィンドウと遺伝子構造ウィンドウの表示
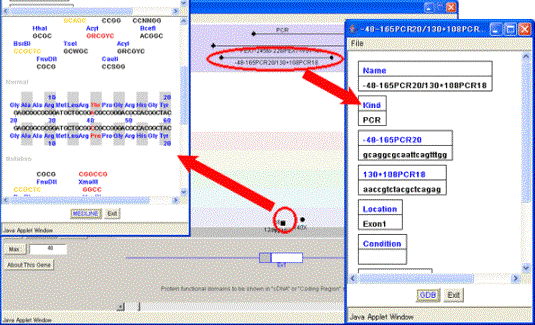
変異 “T14P”変異詳細ウィンドウを表示させ、 “T14P”を挟むプローブ“-48-165PCR20/130+108PCR18”の詳細情報を同時に表示させました。これらの情報を同時に得ることで、変異によって変化する制限酵素と、変異を挟む領域のプライマーを調べることができます。
8.1.12. タンパク質ドメイン構造の表示
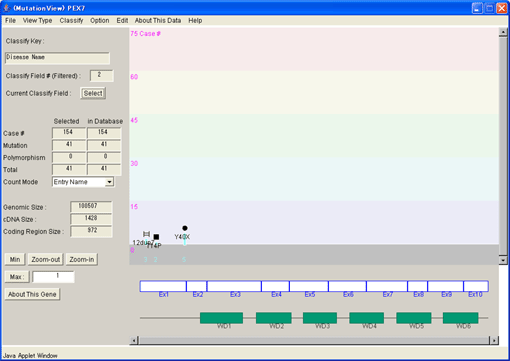
[View Type]メニューで[Coding Region]を選択し、横軸の遺伝子構造を翻訳領域に変更しました。翻訳領域に対応したタンパク質のドメイン構造が表示されています。
8.2. クラシファイ機能の利用2−変異の付加情報を絞り込んで変異を表示する
8.2.1. クラシファイウィンドウの表示
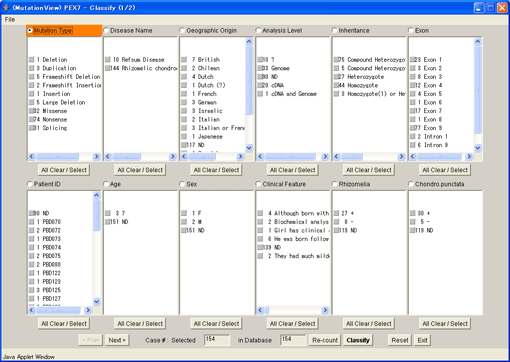
[Classify]メニューを選択し、クラシファイウィンドウを表示させます。
8.2.2. 条件の選択
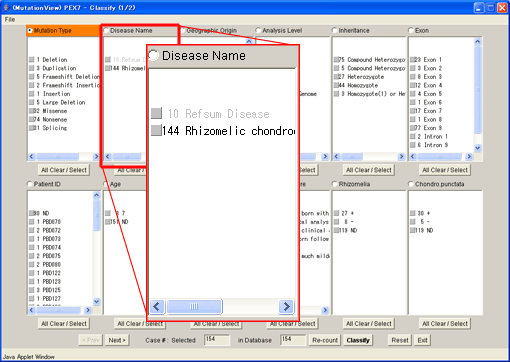
表示させる種別を選択します。“Disease Name”中の“Rhizomelic chondrodysplasia punctata”を選択しました。この選択した状態で[Re-count]ボタンを押します。
8.2.3. 条件に該当する変異数の再表示
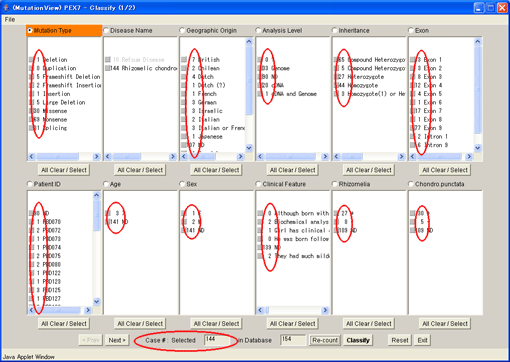
選択された条件で変異数が再計算されて表示されます。それぞれの種別の左に表示されている変異数も再計算されています。
8.2.4. 分類項目の変更
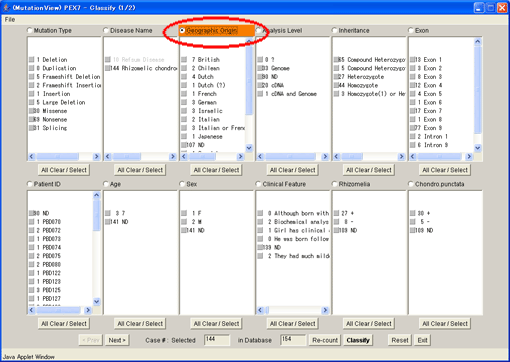
分類項目を“Geographic Origin”に変更しました。
8.2.5. 設定した条件でのクラシファイの実行
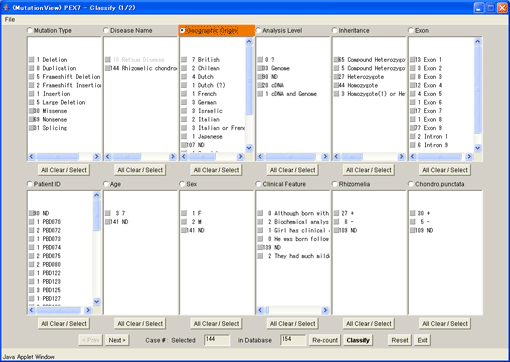
[Classify]ボタンを押し、設定した条件で遺伝子構造ウィンドウを表示させます。
8.2.6. 設定した条件での遺伝子構造ウィンドウの再表示
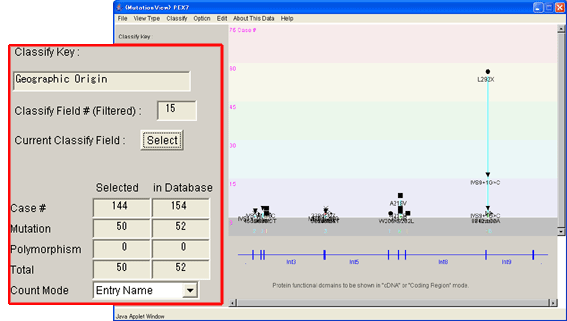
設定した条件で遺伝子構造ウィンドウが再表示されました。[Classify Key]が“Geographic Origin”に変更され、設定された条件での変異数が表示されています。
8.2.7. Zone plotによる変異の表示
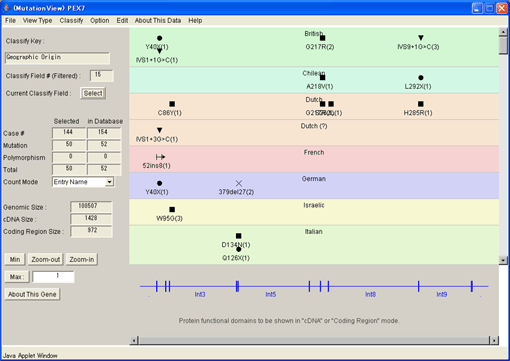
[View Type]メニューで横軸の表示を[Zone plot]に変更しました。“Geographic Origin”に属する種別でゾーンが分けられその中に変異が表示されています。人種における変異の種類と位置のばらつきを見ることができます。
8.2.8. 分類項目中の種別のグループ化
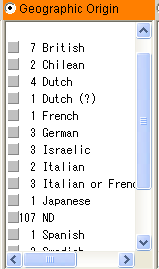
分類項目の種別は予め決められていますが、ユーザーが複数の種別をまとめたグループを作ることができます。クラシファイウィンドウの項目“Geographic Origin”に属する種別をまとめてグループを作る例を挙げます。
8.2.9. グループ化種別の選択
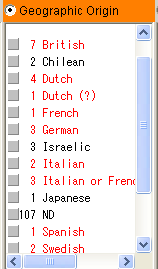
グループ化したい種別を右クリックすると選択されたものは赤色の文字になります。ここではヨーロッパ人のグループを想定し、“British”、“Dutch”などを選択しています。グループ化するものをすべて選択したら[Ctrl]+クリックを押します。
8.2.10. グループ名の入力
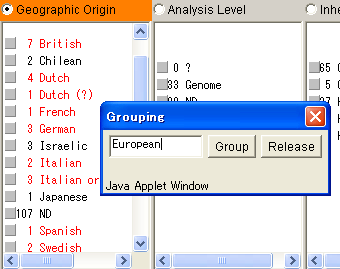
グループ化ウィンドウが表示されました。グループ名を入力し、[Group]ボタンを押します。
8.2.11. グループの決定
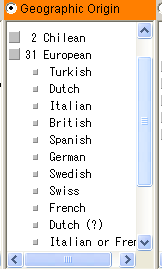
選択された種別をまとめた“European”というグループが作られ、グループに属する種別がグループの下の階層として表示されます。[Classify]ボタンを押してグループ化を遺伝子構造ウィンドウに反映させます。
8.2.12. グループ化後の遺伝子構造ウィンドウの再表示
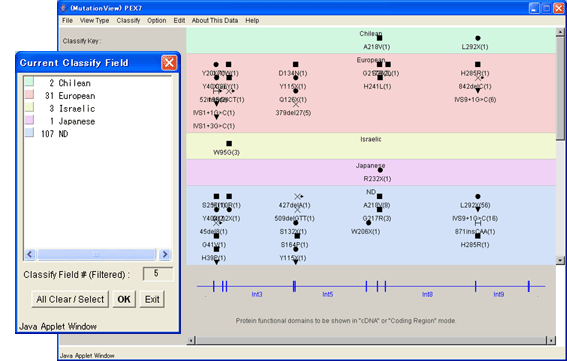
選択された種別に属する変異が新たに作られたグループ“European”のゾーンに表示されています。ツールバーの[Current Classify Field]の[Select]ボタンを押すとグループ“European”が作られています。
9. 発表等
●Minoshima S, Mitsuyama S, Ohtsubo M, Kawamura T, Ito S, Shibamoto S, Ito F, Shimizu N.
The
KMDB/MutationView: a mutation database for human disease genes.
Nucleic Acids Res 2001 Jan 1; 29(1): 327-8
●蓑島伸生,清水信義
遺伝子疾患変異知識データベース MutationViewの構築
医学のあゆみ Vol. 197 No. 13 (2001) 6. 30: 970-978
●蓑島伸生,清水信義
遺伝子疾患知識ベースの構築
蛋白質 核酸 酵素 Vol. 46 No. 16 (2001): 2567-2574
●蓑島伸生,清水信義
疾患遺伝子変異データベース
実験医学 Vol. 19 No. 11 (増刊)2001: 132-140
●蓑島伸生,清水信義
疾患遺伝子変異知識ベースMutationViewバージョン1.2
遺伝子医学 Vol. 5 No. 3 (2001): 140-146
●大坪正史,清水信義,蓑島伸生
バイオ研究者からのバイオNLPへの期待
a)遺伝子変異データベース構築のための情報収集と抽出の現状
情報処理 Vol.46 No.2 (2005): 112-119

●Oxford University Press Bioinformatics Prize(2003)受賞